Actualmente las investigadoras del grupo de Investigación Operativa del DC/ICC trabajan en colaboración con el investigador invitado Carlos Brizuela. A partir de diversos contactos, generados en estos últimos años con nuestra casa de estudios, el objetivo del proyecto es determinar qué clase de proteínas pueden ser diseñadas computacionalmente a través de métodos exactos de optimización.
El doctor Brizuela es investigador titular del Departamento de Ciencias de la Computación del Centro de Investigación Científica y de Educación Superior-CICESE (Baja California, México), donde es fundador y responsable del Laboratorio de Biología Computacional. Sus líneas de investigación abordan el análisis, diseño y aplicación de algoritmos de aprendizaje automático y optimización para problemas en biología computacional, en particular para la identificación y diseño de biomoléculas con potencial terapéutico y de diagnóstico.
Esta nota explora su estancia sabática en Buenos Aires y los principales desafíos que enfrenta su investigación en biología computacional.
En numerosas oportunidades la ciencia está ligada a la interdisciplina, una forma de generar conocimiento que combina enfoques de distintas ciencias para resolver problemas que exceden los límites de una sola disciplina.
Y ese también es el caso de la Bioinformática, campo estratégico de las ciencias computacionales que lleva a cabo el análisis de secuencias de moléculas biológicas y suele procesar grandes volúmenes de información. Este campo puede ayudar a comprender mejor los genes y las proteínas, lo que tiene un impacto muy positivo en la salud así como en otros dominios.
Normalmente se aplica a los genes, al ADN, al ARN, o a las proteínas, y resulta especialmente útil para comparar secuencias de genes y proteínas entre distintos organismos, pudiendo ver las relaciones evolutivas entre organismos, e intentando averiguar cuál es la función de dichos genes y proteínas. Para entenderlo con una analogía: la bioinformática se encarga de la parte “lingüística” de la genética. De la misma manera que los lingüistas estudian los patrones en el lenguaje, los bioinformáticos estudian los patrones en las secuencias de ADN o de proteínas.
Pero, ¿qué pasaría si, además, estos problemas propios de la bioinformática se combinan con la investigación operativa, particularmente con la optimización exacta?
En esta historia aparece el investigador Carlos Brizuela -responsable del Laboratorio de Biología Computacional del Departamento de Ciencias de la Computación del CICESE, México- quien este año decidió tomarse un sabático en Buenos Aires, más precisamente en el Instituto de Ciencias de la Computación, con sede en Exactas.
“Conocí a las doctoras Irene Loiseau y Paula Zabala, por diversas colaboraciones que tenemos desde hace años. Recientemente también me contacté con la doctora Isabel Méndez Díaz, ya que en su grupo tienen un manejo muy avanzado de la investigación operativa. Así empezamos a colaborar en un problema de diseño y modelado de proteínas que estamos abordando hoy en el CICESE”, puntualiza Brizuela.
El problema principal radica en el diseño computacional de proteínas y el desafío es modelarlo como un problema de optimización exacta. El doctor Brizuela, quien también trabaja en el proyecto con Carlos Oliva (Becario de CICESE, en estancia en el ICC), está indagando si la proteína real es el óptimo de un problema de optimización que se está modelando hoy en día y determina si la solución biológica coincide con la solución computacional.
Estas proteínas son globulares, sin ninguna función en particular. Y en el caso de la tesis de doctorado de Oliva, son péptidos cíclicos, proteínas muy pequeñas que cierran un ciclo y poseen un interés terapéutico considerable porque es más difícil que se degraden, ya que son más estables.
“Desde que se planteó el diseño de proteínas a fines de los años 80, este ha sido modelado como un problema de optimización. En este caso la proteína adopta su conformación de mínima energía, sino fuera así la conformación sería inestable. Si uno pudiese modelar esta función de energía y encontrar cuál es la configuración que la minimiza, sabría exactamente la forma de la proteína. Y el diseño es el problema inverso en el cual tenemos la forma y queremos saber cuál es la cadena de aminoácidos que cuando se pliega en su configuración de mínima energía va a adoptar esa forma”, explica Brizuela.
El investigador comenta que con su equipo del CICESE, y junto a la colaboración que está realizando en el ICC, está demostrando que las funciones de energía que se plantean actualmente no modelan a la biología como tal. “Demostramos que la solución biológica no coincide con el óptimo, es decir que la solución biológica bajo esta función de optimización es de peor calidad que el óptimo de la función que modela al problema. Esto significa que tenemos que encontrar nuevas funciones de energía, que modelen mejor a la biología”, aclara.
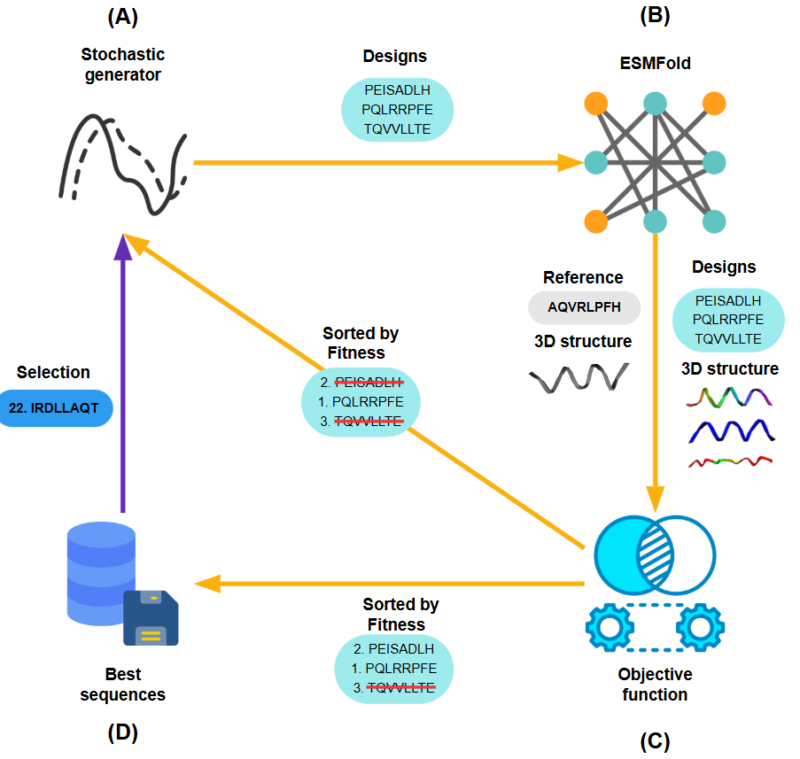
Figura 1 – Esquema del procedimiento de diseño propuesto.
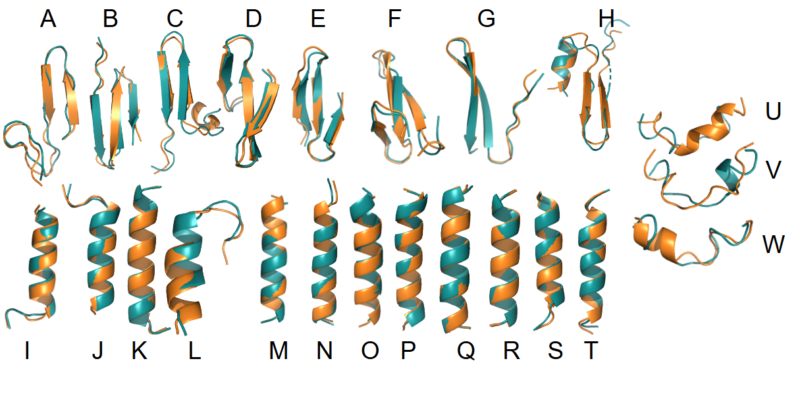
Figura 2 – Resultado del diseño donde se superponen la proteína objetivo de diseño (dorada) con la proteína diseñada (verde).
Al mismo tiempo, otro proyecto muy relevante que lidera Brizuela es el diseño de nuevos antibióticos basados en péptidos antimicrobianos, proteínas de tamaño pequeño capaces de matar a bacterias actualmente resistentes a varios antibióticos comerciales que producen infecciones en humanos (una de las principales causas de muerte estimadas para 2050).
“Presentan una diferencia enorme con los antibióticos tradicionales que se basan en moléculas pequeñas de hasta decenas de átomos. Estos péptidos surgieron en la década de los 80, son pequeñas cadenas de aminoácidos normalmente de 8 hasta 30 aminoácidos aproximadamente y se descubrió que muchos de ellos son parte del sistema inmune innato, los generamos para matar bacterias y hay una cantidad enorme en nuestro cuerpo”, describe el investigador.
En este caso el investigador busca diseñar modelos para uno de estos antibióticos, que logren “matar” con mayor eficiencia una bacteria resistente. Sin dudas, este tipo de fármacos ofrecen muchas ventajas como sus amplios mecanismos de acción y el producto después de su degradación se convierte en aminoácidos que pueden ser reciclados para su uso por el organismo.
Desafíos de investigación en biología computacional del CICESE
El Centro de Investigación Científica y de Educación Superior (CICESE) es una institución de referencia científica que está ubicada en la ciudad de Ensenada, estado de Baja California, México. Pertenece al sistema de centros públicos de investigación del Consejo Nacional de Ciencia, Humanidades y Tecnología (CONAHCyT), fue fundado en 1973 y a lo largo de más de cinco décadas, ha evolucionado hasta convertirse en uno de los principales centros científicos de México. Surgió por la necesidad inicial de contar con investigaciones sobre el mar y las ciencias de la tierra, ya que está muy cercano a una zona sísmica, la Falla de San Andrés, por lo que tiene un compromiso histórico con estos tópicos.
Sus investigadores, técnicos y estudiantes de posgrado integran cuatro divisiones muy específicas: Física aplicada, Biología experimental y aplicada, Oceanología y Ciencias de la tierra. En particular, el doctor Brizuela dirige el Laboratorio de Biología Computacional dependiente del Departamento de Ciencias de la Computación, división de Física Aplicada. Su trabajo de investigación es 100% multidisciplinario, ya que colabora permanentemente con el Departamento de Innovación Biomédica que depende de la división de Biología experimental y aplicada.
“Hacer converger las necesidades de investigación que tienen los investigadores del área biomédica con las necesidades de ingenieros y científicos de la computación, es un tremendo desafío”, expresa Brizuela. En este sentido, el investigador comenta que su equipo se ocupa de desarrollar los diseños y encontrar métodos algorítmicos cada vez más eficientes, mientras que los investigadores biomédicos testean esos diseños en el laboratorio, ya sea modelos in vitro o en ratones.
“Lo que le interesa a la contraparte no son tanto los métodos para diseñar proteínas sino el diseño como un producto aplicado a un problema específico. Por ejemplo, buscan nuevos fármacos para algún tipo de cáncer, tienen un target, es decir, una proteína a la cual se le debe acoplar un fármaco o alguna otra proteína para inhibirla. Entonces ellos se enfocan estrictamente en el diseño, no les interesa tanto si el método mejoró un 10 % pero aún no se resuelve el problema”, afirma el investigador del CICESE.
No obstante, para el equipo liderado por Brizuela y para la comunidad científica en computación mejorar al menos un 10% el método de diseño es un gran avance. “Tenemos esa tensión, entendible por cierto, entre la urgencia del área biomédica por atacar un problema o una enfermedad específica y el proceso natural de mejorar la investigación y el método computacional. Lo que aprendí en estos años es que más que tratar de convencer a la contraparte de que esto es un proceso lento, necesitamos buscar a la contraparte que entiende la dinámica del proceso y tiene la apertura mental suficiente para administrar las expectativas de lograr una solución rápida”, concluye.
La vida académica en Exactas-UBA
Brizuela comenta que le encanta trabajar en Buenos Aires, y en particular está fascinado por la vida académica y el ambiente que se respira en la Facultad de Ciencias Exactas y Naturales. “Tener a investigadores de casi todas las ciencias exactas y naturales al alcance de la mano es algo maravilloso, es el ámbito ideal para la colaboración multi, inter y trans-disciplinaria. En el ICC estamos explorando también posibilidades de colaboración con otros investigadores, dada la riqueza de especialidades que manejan aquí. Además, puedo caminar hacia el Pabellón 2 y conversar con investigadores de Biología o Química que trabajan en temas tan fascinantes como el fenómeno conocido como ‘frustración en proteínas’ o con físicos del Pabellón 1 expertos en dinámica de péptidos. De hecho, hoy mismo concretamos una reunión con investigadores que trabajan en la identificación de biomarcadores para el diagnóstico en tuberculosis, ya que otro de los temas que nos interesa es diseñar nuevos marcadores en tuberculosis, una enfermedad en constante crecimiento en México”, comenta entusiasmado.
Doctor Carlos Brizuela Rodríguez
Carlos Brizuela Rodríguez es originario de Asunción, Paraguay, y reside en Ensenada, México. Es Técnico Superior en Electrónica por la Universidad Nacional de Asunción e Ingeniero Industrial en Electrónica, por el Instituto Tecnológico de Tijuana. Tiene una Maestría en Ciencias en Electrónica y Telecomunicaciones por el CICESE y el grado de Ph.D. en Ciencias de la Información y Producción por la Universidad de Tecnología de Kioto. Posee entrenamiento en Mecatrónica en Osaka y en Electrónica Industrial en Taiwan. Fue profesor visitante por un año en el Laboratorio de Descubrimiento computacional de Fármacos en la Skaggs School of Pharmacy and Pharmaceutical Sciences de la Universidad de California en San Diego.
En el 2008 participó en la creación del primer Doctorado en Ingeniería Informática del Paraguay co-dirigiendo a sus primeros dos egresados. Es investigador titular del Departamento de Ciencias de la Computación del CICESE donde es fundador y responsable del laboratorio de Biología Computacional. Sus líneas de investigación están vinculadas al análisis, diseño y aplicación de algoritmos de aprendizaje automático y optimización para problemas en biología computacional, en particular para la identificación y diseño de biomoléculas con potencial farmacológico y de diagnóstico. Ha graduado 40 estudiantes de maestría y 16 de doctorado, ha sido evaluador en más de 130 tesis de maestría y doctorado de los Programas Nacionales de Posgrados de Calidad y del extranjero. Ha publicado más de 90 artículos de investigación entre revistas del JCR y congresos internacionales. Es miembro del Sistema Nacional de Investigadores de México desde el 2002.
Actualmente está en estancia sabática en el Instituto de Ciencias de la Computación-UBA/CONICET (marzo 2024-febrero 2025).